七、线性回归和健康结果
糖尿病数据集
我们将使用来自糖尿病患者的数据集。 数据由 442 个样本和 10 个变量(都是生理特征)组成,因此它很高而且很窄。 因变量是基线后一年疾病进展的定量测量。
这是一个经典的数据集,由 Efron,Hastie,Johnstone 和 Tibshirani 在他们的最小角度回归的论文中使用,也是 scikit-learn 中包含的众多数据集之一。
data = datasets.load_diabetes()
feature_names=['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6']
trn,test,y_trn,y_test = train_test_split(data.data, data.target, test_size=0.2)
trn.shape, test.shape
# ((353, 10), (89, 10))
Sklearn 中的线性回归
考虑系统Xβ=y,其中X的行比列更多。 当你有比变量更多的数据样本时会发生这种情况。 我们想要找到 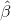 来最小化:
来最小化:

让我们从使用 sklearn 实现开始:
regr = linear_model.LinearRegression()
%timeit regr.fit(trn, y_trn)
# 458 µs ± 62.4 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
pred = regr.predict(test)
有一些指标来表示我们的预测有多好,会很有帮助。 我们将研究均方范数(L2)和平均绝对误差(L1)。
def regr_metrics(act, pred):
return (math.sqrt(metrics.mean_squared_error(act, pred)),
metrics.mean_absolute_error(act, pred))
regr_metrics(y_test, regr.predict(test))
# (75.36166834955054, 60.629082113104403)
多项式特征
线性回归找到最佳系数βi:

添加多项式特征仍然是线性回归问题,只需更多项:
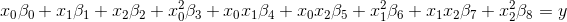
我们需要使用原始数据X来计算其他多项式特征。
trn.shape
# (353, 10)
现在,我们想通过添加更多功能,来尝试提高模型的表现。 目前,我们的模型在每个变量中都是线性的,但我们可以添加多项式特征来改变它。
poly = PolynomialFeatures(include_bias=False)
trn_feat = poly.fit_transform(trn)
', '.join(poly.get_feature_names(feature_names))
# 'age, sex, bmi, bp, s1, s2, s3, s4, s5, s6, age^2, age sex, age bmi, age bp, age s1, age s2, age s3, age s4, age s5, age s6, sex^2, sex bmi, sex bp, sex s1, sex s2, sex s3, sex s4, sex s5, sex s6, bmi^2, bmi bp, bmi s1, bmi s2, bmi s3, bmi s4, bmi s5, bmi s6, bp^2, bp s1, bp s2, bp s3, bp s4, bp s5, bp s6, s1^2, s1 s2, s1 s3, s1 s4, s1 s5, s1 s6, s2^2, s2 s3, s2 s4, s2 s5, s2 s6, s3^2, s3 s4, s3 s5, s3 s6, s4^2, s4 s5, s4 s6, s5^2, s5 s6, s6^2'
trn_feat.shape
# (353, 65)
regr.fit(trn_feat, y_trn)
# LinearRegression(copy_X=True, fit_intercept=True, n_jobs=1, normalize=False)
regr_metrics(y_test, regr.predict(poly.fit_transform(test)))
# (55.747345922929185, 42.836164292252235)
时间对于特征数是平方的,对于样本数是线性的,所以这将变得非常慢!
%timeit poly.fit_transform(trn)
# 635 µs ± 9.25 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
加速特征生成
我们想加快速度。 我们将使用 Numba,一个直接将代码编译为 C 的 Python 库。
Numba 是一个编译器。
资源
Jake VanderPlas 的这个教程是一个很好的介绍。 在这里,Jake 使用 Numba 实现了一个非平凡的算法(非均匀快速傅里叶变换)。
Cython 是另一种选择。 我发现 Cython 主要比 Numba 更多的知识(它更接近 C),但提供类似 Numba 的加速。

这里是预先编译(AOT)编译器,即时编译(JIT)编译器和解释器之间差异的全面回答。
使用向量化和原生代码进行实验
让我们先了解一下 Numba,然后我们将回到我们的糖尿病数据集回归的多项式特征问题。
%matplotlib inline
import math, numpy as np, matplotlib.pyplot as plt
from pandas_summary import DataFrameSummary
from scipy import ndimage
from numba import jit, vectorize, guvectorize, cuda, float32, void, float64
我们将展示以下方面的影响:
- 避免内存分配和副本(比 CPU 计算慢)
- 更好的局部性
- 向量化
如果我们一次在整个数组上使用 numpy,它会创建大量的临时值,并且不能使用缓存。 如果我们一次使用 numba 循环遍历数组项,那么我们就不必分配大型临时数组,并且可以复用缓存数据,因为我们正在对每个数组项进行多次计算。
# 无类型和没有向量化
def proc_python(xx,yy):
zz = np.zeros(nobs, dtype='float32')
for j in range(nobs):
x, y = xx[j], yy[j]
x = x*2 - ( y * 55 )
y = x + y*2
z = x + y + 99
z = z * ( z - .88 )
zz[j] = z
return zz
nobs = 10000
x = np.random.randn(nobs).astype('float32')
y = np.random.randn(nobs).astype('float32')
%timeit proc_python(x,y)
# 49.8 ms ± 1.19 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
NumPy
Numpy 让我们对其向量化:
# 有类型和向量化
def proc_numpy(x,y):
z = np.zeros(nobs, dtype='float32')
x = x*2 - ( y * 55 )
y = x + y*2
z = x + y + 99
z = z * ( z - .88 )
return z
np.allclose( proc_numpy(x,y), proc_python(x,y), atol=1e-4 )
# True
%timeit proc_numpy(x,y) # Typed and vectorized
# 35.9 µs ± 166 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
Numba
Numba 提供几种不同的装饰器。 我们将尝试两种不同的方法:
@jit:非常一般@vectorize:不需要编写for循环。操作相同大小的向量时很有用
首先,我们将使用 Numba 的jit(即时)编译器装饰器,而无需显式向量化。 这避免了大量内存分配,因此我们有更好的局部性:
@jit()
def proc_numba(xx,yy,zz):
for j in range(nobs):
x, y = xx[j], yy[j]
x = x*2 - ( y * 55 )
y = x + y*2
z = x + y + 99
z = z * ( z - .88 )
zz[j] = z
return zz
z = np.zeros(nobs).astype('float32')
np.allclose( proc_numpy(x,y), proc_numba(x,y,z), atol=1e-4 )
# True
%timeit proc_numba(x,y,z)
# 6.4 µs ± 17.6 ns per loop (mean ± std. dev. of 7 runs, 100000 loops each)
现在我们将使用 Numba 的vectorize装饰器。 Numba 的编译器以比普通 Python 和 Numpy 更聪明的方式优化它。 它为你写了一个 Numpy ufunc,传统上它涉及编写 C 并且不那么简单。
@vectorize
def vec_numba(x,y):
x = x*2 - ( y * 55 )
y = x + y*2
z = x + y + 99
return z * ( z - .88 )
np.allclose(vec_numba(x,y), proc_numba(x,y,z), atol=1e-4 )
# True
%timeit vec_numba(x,y)
# 5.82 µs ± 14.4 ns per loop (mean ± std. dev. of 7 runs, 100000 loops each)
Numba 很棒。 看看这有多快!
Numba 多项式特征
@jit(nopython=True)
def vec_poly(x, res):
m,n=x.shape
feat_idx=0
for i in range(n):
v1=x[:,i]
for k in range(m): res[k,feat_idx] = v1[k]
feat_idx+=1
for j in range(i,n):
for k in range(m): res[k,feat_idx] = v1[k]*x[k,j]
feat_idx+=1
行序和列序存储
“矩阵的行序布局将第一行放在连续的内存中,然后是第二行放在它后面,然后是第三行,依此类推。列序布局将第一列放在连续内存中,然后放入第二列,等等....虽然知道特定数据集使用哪种布局对于良好的性能至关重要,但对于哪种布局“更好”的问题,没有单一的答案。”
“事实证明,匹配算法与数据布局的工作方式,可以决定应用程序的性能。”
“简短的说法是:始终按照布局顺序遍历数据。”
列序布局:Fortran,Matlab,R 和 Julia
行序布局:C,C ++,Python,Pascal,Mathematica
trn = np.asfortranarray(trn)
test = np.asfortranarray(test)
m,n=trn.shape
n_feat = n*(n+1)//2 + n
trn_feat = np.zeros((m,n_feat), order='F')
test_feat = np.zeros((len(y_test), n_feat), order='F')
vec_poly(trn, trn_feat)
vec_poly(test, test_feat)
regr.fit(trn_feat, y_trn)
# LinearRegression(copy_X=True, fit_intercept=True, n_jobs=1, normalize=False)
regr_metrics(y_test, regr.predict(test_feat))
# (55.74734592292935, 42.836164292252306)
%timeit vec_poly(trn, trn_feat)
# 7.33 µs ± 19.8 ns per loop (mean ± std. dev. of 7 runs, 100000 loops each)
回想一下,这是 sklearn PolynomialFeatures实现的时间,它是由专家创建的:
%timeit poly.fit_transform(trn)
# 635 µs ± 9.25 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
605/7.7
# 78.57142857142857
这是一个大问题! Numba 太神奇了! 只需一行代码,我们就可以获得比 scikit 学习快 78 倍的速度(由专家优化)。
正则化和噪声
正则化是一种减少过拟合,并创建更好地泛化到新数据的模型的方法。
正则化
Lasso 回归使用 L1 惩罚,产生稀疏系数。 参数α用于加权惩罚项。 Scikit Learn 的LassoCV使用许多不同的α值进行交叉验证。
观看 Lasso 回归的 Coursera 视频,了解更多信息。
reg_regr = linear_model.LassoCV(n_alphas=10)
reg_regr.fit(trn_feat, y_trn)
'''
/home/jhoward/anaconda3/lib/python3.6/site-packages/sklearn/linear_model/coordinate_descent.py:484: ConvergenceWarning: Objective did not converge. You might want to increase the number of iterations. Fitting data with very small alpha may cause precision problems.
ConvergenceWarning)
LassoCV(alphas=None, copy_X=True, cv=None, eps=0.001, fit_intercept=True,
max_iter=1000, n_alphas=10, n_jobs=1, normalize=False, positive=False,
precompute='auto', random_state=None, selection='cyclic', tol=0.0001,
verbose=False)
'''
reg_regr.alpha_
# 0.0098199431661591518
regr_metrics(y_test, reg_regr.predict(test_feat))
# (50.0982471642817, 40.065199085003101)
噪声
现在我们将为数据添加一些噪音。
idxs = np.random.randint(0, len(trn), 10)
y_trn2 = np.copy(y_trn)
y_trn2[idxs] *= 10 # label noise
regr = linear_model.LinearRegression()
regr.fit(trn, y_trn)
regr_metrics(y_test, regr.predict(test))
# (51.1766253181518, 41.415992803872754)
regr.fit(trn, y_trn2)
regr_metrics(y_test, regr.predict(test))
# (62.66110319520415, 53.21914420254862)
Huber 损失是一种损失函数,对异常值的敏感度低于平方误差损失。 对于小的误差值,它是二次的,对于大的值,它是线性的。
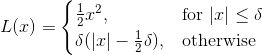
hregr = linear_model.HuberRegressor()
hregr.fit(trn, y_trn2)
regr_metrics(y_test, hregr.predict(test))
# (51.24055602541746, 41.670840571376822)