八、如何实现线性回归
在上一课中,我们使用 scikit learn 的实现计算了糖尿病数据集的最小二乘线性回归。 今天,我们将看看如何编写自己的实现。
起步
from sklearn import datasets, linear_model, metrics
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import PolynomialFeatures
import math, scipy, numpy as np
from scipy import linalg
np.set_printoptions(precision=6)
data = datasets.load_diabetes()
feature_names=['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6']
trn,test,y_trn,y_test = train_test_split(data.data, data.target, test_size=0.2)
trn.shape, test.shape
# ((353, 10), (89, 10))
def regr_metrics(act, pred):
return (math.sqrt(metrics.mean_squared_error(act, pred)),
metrics.mean_absolute_error(act, pred))
sklearn 如何实现它
sklearn 是如何做到这一点的? 通过检查源代码,你可以看到在密集的情况下,它调用scipy.linalg.lstqr,它调用 LAPACK 方法:
选项是
'gelsd','gelsy','gelss'。默认值'gelsd'是个好的选择,但是,'gelsy'在许多问题上更快一些。'gelss'由于历史原因而使用。它通常更慢但是使用更少内存。
Scipy 稀疏最小二乘
我们不会详细介绍稀疏版本的最小二乘法。如果你有兴趣,请参考以下信息:
Scipy 稀疏最小二乘使用称为 Golub 和 Kahan 双对角化的迭代方法。
Scipy 稀疏最小二乘源代码:预处理是减少迭代次数的另一种方法。如果有可能有效地求解相关系统M*x = b,其中M以某种有用的方式近似A(例如,M-A具有低秩或其元素相对于A的元素较小),则 LSQR 可以在系统A*M(inverse)*z = b更快地收敛。之后可以通过求解M * x = z来恢复x。
如果A是对称的,则不应使用 LSQR!替代方案是对称共轭梯度法(cg)和/或 SYMMLQ。 SYMMLQ 是对称 cg 的一种实现,适用于任何对称的A,并且比 LSQR 更快收敛。如果A是正定的,则存在对称 cg 的其他实现,每次迭代需要的工作量比 SYMMLQ 少一些(但需要相同的迭代次数)。
linalg.lstqr
sklearn 实现为我们添加一个常数项(因为对于我们正在学习的直线,y截距可能不是 0)。 我们现在需要手工完成:
trn_int = np.c_[trn, np.ones(trn.shape[0])]
test_int = np.c_[test, np.ones(test.shape[0])]
由于linalg.lstsq允许我们指定,我们想要使用哪个 LAPACK 例程,让我们尝试它们并进行一些时间比较:
%timeit coef, _,_,_ = linalg.lstsq(trn_int, y_trn, lapack_driver="gelsd")
# 290 µs ± 9.24 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
%timeit coef, _,_,_ = linalg.lstsq(trn_int, y_trn, lapack_driver="gelsy")
# 140 µs ± 91.7 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
%timeit coef, _,_,_ = linalg.lstsq(trn_int, y_trn, lapack_driver="gelss")
# 199 µs ± 228 ns per loop (mean ± std. dev. of 7 runs, 1000 loops each)
朴素解法
回想一下,我们想找到 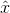 ,来最小化:
,来最小化:

另一种思考方式是,我们对向量b最接近A的子空间(称为A的范围)的地方感兴趣。 这是b在A上的投影。由于  必须垂直于
必须垂直于A的子空间,我们可以看到:

使用了  因为要相乘
因为要相乘A和  的每列。
的每列。
这让我们得到正规方程:
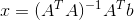
def ls_naive(A, b):
return np.linalg.inv(A.T @ A) @ A.T @ b
%timeit coeffs_naive = ls_naive(trn_int, y_trn)
# 45.8 µs ± 4.65 µs per loop (mean ± std. dev. of 7 runs, 10000 loops each)
coeffs_naive = ls_naive(trn_int, y_trn)
regr_metrics(y_test, test_int @ coeffs_naive)
# (57.94102134545707, 48.053565198516438)
正规方程(Cholesky)
正规方程:

如果A具有满秩,则伪逆  是正方形,埃尔米特正定矩阵。 解决这种系统的标准方法是 Cholesky 分解,它找到上三角形
是正方形,埃尔米特正定矩阵。 解决这种系统的标准方法是 Cholesky 分解,它找到上三角形R,满足  。
。
以下步骤基于 Trefethen 的算法 11.1:
A = trn_int
b = y_trn
AtA = A.T @ A
Atb = A.T @ b
警告:对于Cholesky,Numpy 和 Scipy 默认为不同的上/下三角。
R = scipy.linalg.cholesky(AtA)
np.set_printoptions(suppress=True, precision=4)
R
'''
array([[ 0.9124, 0.1438, 0.1511, 0.3002, 0.2228, 0.188 ,
-0.051 , 0.1746, 0.22 , 0.2768, -0.2583],
[ 0. , 0.8832, 0.0507, 0.1826, -0.0251, 0.0928,
-0.3842, 0.2999, 0.0911, 0.15 , 0.4393],
[ 0. , 0. , 0.8672, 0.2845, 0.2096, 0.2153,
-0.2695, 0.3181, 0.3387, 0.2894, -0.005 ],
[ 0. , 0. , 0. , 0.7678, 0.0762, -0.0077,
0.0383, 0.0014, 0.165 , 0.166 , 0.0234],
[ 0. , 0. , 0. , 0. , 0.8288, 0.7381,
0.1145, 0.4067, 0.3494, 0.158 , -0.2826],
[ 0. , 0. , 0. , 0. , 0. , 0.3735,
-0.3891, 0.2492, -0.3245, -0.0323, -0.1137],
[ 0. , 0. , 0. , 0. , 0. , 0. ,
0.6406, -0.511 , -0.5234, -0.172 , -0.9392],
[ 0. , 0. , 0. , 0. , 0. , 0. ,
0. , 0.2887, -0.0267, -0.0062, 0.0643],
[ 0. , 0. , 0. , 0. , 0. , 0. ,
0. , 0. , 0.2823, 0.0636, 0.9355],
[ 0. , 0. , 0. , 0. , 0. , 0. ,
0. , 0. , 0. , 0.7238, 0.0202],
[ 0. , 0. , 0. , 0. , 0. , 0. ,
0. , 0. , 0. , 0. , 18.7319]])
'''
检查我们的分解:
np.linalg.norm(AtA - R.T @ R)
# 4.5140158187158533e-16

w = scipy.linalg.solve_triangular(R, Atb, lower=False, trans='T')
检查我们的结果是否符合预期总是好的:(以防我们输入错误的参数,函数没有返回我们想要的东西,或者有时文档甚至过时)。
np.linalg.norm(R.T @ w - Atb)
# 1.1368683772161603e-13
coeffs_chol = scipy.linalg.solve_triangular(R, w, lower=False)
np.linalg.norm(R @ coeffs_chol - w)
# 6.861429794408013e-14
def ls_chol(A, b):
R = scipy.linalg.cholesky(A.T @ A)
w = scipy.linalg.solve_triangular(R, A.T @ b, trans='T')
return scipy.linalg.solve_triangular(R, w)
%timeit coeffs_chol = ls_chol(trn_int, y_trn)
# 111 µs ± 272 ns per loop (mean ± std. dev. of 7 runs, 10000 loops each)
coeffs_chol = ls_chol(trn_int, y_trn)
regr_metrics(y_test, test_int @ coeffs_chol)
# (57.9410213454571, 48.053565198516438)
QR 分解

def ls_qr(A,b):
Q, R = scipy.linalg.qr(A, mode='economic')
return scipy.linalg.solve_triangular(R, Q.T @ b)
%timeit coeffs_qr = ls_qr(trn_int, y_trn)
# 205 µs ± 264 ns per loop (mean ± std. dev. of 7 runs, 1000 loops each)
coeffs_qr = ls_qr(trn_int, y_trn)
regr_metrics(y_test, test_int @ coeffs_qr)
# (57.94102134545711, 48.053565198516452)
SVD

SVD 给出伪逆。
def ls_svd(A,b):
m, n = A.shape
U, sigma, Vh = scipy.linalg.svd(A, full_matrices=False)
w = (U.T @ b)/ sigma
return Vh.T @ w
%timeit coeffs_svd = ls_svd(trn_int, y_trn)
# 1.11 ms ± 320 ns per loop (mean ± std. dev. of 7 runs, 1000 loops each)
%timeit coeffs_svd = ls_svd(trn_int, y_trn)
# 266 µs ± 8.49 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
coeffs_svd = ls_svd(trn_int, y_trn)
regr_metrics(y_test, test_int @ coeffs_svd)
# (57.941021345457244, 48.053565198516687)
最小二乘回归的随机 Sketching 技巧
线性 Sketching(Woodruff)
- 抽取
r×n随机矩阵S,r << n - 计算
S A和S b - 找到回归
SA x = Sb的精确解x
时间比较
import timeit
import pandas as pd
def scipylstq(A, b):
return scipy.linalg.lstsq(A,b)[0]
row_names = ['Normal Eqns- Naive',
'Normal Eqns- Cholesky',
'QR Factorization',
'SVD',
'Scipy lstsq']
name2func = {'Normal Eqns- Naive': 'ls_naive',
'Normal Eqns- Cholesky': 'ls_chol',
'QR Factorization': 'ls_qr',
'SVD': 'ls_svd',
'Scipy lstsq': 'scipylstq'}
m_array = np.array([100, 1000, 10000])
n_array = np.array([20, 100, 1000])
index = pd.MultiIndex.from_product([m_array, n_array], names=['# rows', '# cols'])
pd.options.display.float_format = '{:,.6f}'.format
df = pd.DataFrame(index=row_names, columns=index)
df_error = pd.DataFrame(index=row_names, columns=index)
# %%prun
for m in m_array:
for n in n_array:
if m >= n:
x = np.random.uniform(-10,10,n)
A = np.random.uniform(-40,40,[m,n]) # removed np.asfortranarray
b = np.matmul(A, x) + np.random.normal(0,2,m)
for name in row_names:
fcn = name2func[name]
t = timeit.timeit(fcn + '(A,b)', number=5, globals=globals())
df.set_value(name, (m,n), t)
coeffs = locals()[fcn](A, b)
reg_met = regr_metrics(b, A @ coeffs)
df_error.set_value(name, (m,n), reg_met[0])
df
| # rows | 100 | 1000 | 10000 | ||||||
|---|---|---|---|---|---|---|---|---|---|
| # cols | 20 | 100 | 1000 | 20 | 100 | 1000 | 20 | 100 | 1000 |
| Normal Eqns- Naive | 0.001276 | 0.003634 | NaN | 0.000960 | 0.005172 | 0.293126 | 0.002226 | 0.021248 | 1.164655 |
| Normal Eqns- Cholesky | 0.001660 | 0.003958 | NaN | 0.001665 | 0.004007 | 0.093696 | 0.001928 | 0.010456 | 0.399464 |
| QR Factorization | 0.002174 | 0.006486 | NaN | 0.004235 | 0.017773 | 0.213232 | 0.019229 | 0.116122 | 2.208129 |
| SVD | 0.003880 | 0.021737 | NaN | 0.004672 | 0.026950 | 1.280490 | 0.018138 | 0.130652 | 3.433003 |
| Scipy lstsq | 0.004338 | 0.020198 | NaN | 0.004320 | 0.021199 | 1.083804 | 0.012200 | 0.088467 | 2.134780 |
df_error
| # rows | 100 | 1000 | 10000 | ||||||
|---|---|---|---|---|---|---|---|---|---|
| # cols | 20 | 100 | 1000 | 20 | 100 | 1000 | 20 | 100 | 1000 |
| Normal Eqns- Naive | 1.702742 | 0.000000 | NaN | 1.970767 | 1.904873 | 0.000000 | 1.978383 | 1.980449 | 1.884440 |
| Normal Eqns- Cholesky | 1.702742 | 0.000000 | NaN | 1.970767 | 1.904873 | 0.000000 | 1.978383 | 1.980449 | 1.884440 |
| QR Factorization | 1.702742 | 0.000000 | NaN | 1.970767 | 1.904873 | 0.000000 | 1.978383 | 1.980449 | 1.884440 |
| SVD | 1.702742 | 0.000000 | NaN | 1.970767 | 1.904873 | 0.000000 | 1.978383 | 1.980449 | 1.884440 |
| Scipy lstsq | 1.702742 | 0.000000 | NaN | 1.970767 | 1.904873 | 0.000000 | 1.978383 | 1.980449 | 1.884440 |
store = pd.HDFStore('least_squares_results.h5')
store['df'] = df
'''
C:\Users\rache\Anaconda3\lib\site-packages\IPython\core\interactiveshell.py:2881: PerformanceWarning:
your performance may suffer as PyTables will pickle object types that it cannot
map directly to c-types [inferred_type->floating,key->block0_values] [items->[(100, 20), (100, 100), (100, 1000), (1000, 20), (1000, 100), (1000, 1000), (5000, 20), (5000, 100), (5000, 1000)]]
exec(code_obj, self.user_global_ns, self.user_ns)
'''
注解
我用魔术指令%prun来测量我的代码。
替代方案:最小绝对偏差(L1 回归)
- 异常值的敏感度低于最小二乘法。
- 没有闭式解,但可以通过线性规划解决。
条件作用和稳定性
条件数
条件数是一个指标,衡量输入的小变化导致输出变化的程度。
问题:为什么我们在数值线性代数中,关心输入的小变化的相关行为?
相对条件数由下式定义:
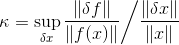
其中  是无穷小。
是无穷小。
根据 Trefethen(第 91 页),如果κ很小(例如 1, 10, 10^2),问题是良态的,如果κ很大(例如10^6, 10^16),那么问题是病态的。
条件作用:数学问题的扰动行为(例如最小二乘)
稳定性:用于在计算机上解决该问题的算法的扰动行为(例如,最小二乘算法,householder,回代,高斯消除)
条件作用的例子
计算非对称矩阵的特征值的问题通常是病态的。
A = [[1, 1000], [0, 1]]
B = [[1, 1000], [0.001, 1]]
wA, vrA = scipy.linalg.eig(A)
wB, vrB = scipy.linalg.eig(B)
wA, wB
'''
(array([ 1.+0.j, 1.+0.j]),
array([ 2.00000000e+00+0.j, -2.22044605e-16+0.j]))
'''
矩阵的条件数
乘积  经常出现,它有自己的名字:
经常出现,它有自己的名字:A的条件数。注意,通常我们谈论问题的条件作用,而不是矩阵。
A的条件数涉及:
- 给定
Ax = b中的A和x,计算b - 给定
Ax = b中的A和b,计算x
未交待清楚的事情
完整和简化分解
SVD
来自 Trefethen 的图:
对于所有矩阵,QR 分解都存在
与 SVD 一样,有 QR 分解的完整版和简化版。
矩阵的逆是不稳定的
from scipy.linalg import hilbert
n = 5
hilbert(n)
'''
array([[ 1. , 0.5 , 0.3333, 0.25 , 0.2 ],
[ 0.5 , 0.3333, 0.25 , 0.2 , 0.1667],
[ 0.3333, 0.25 , 0.2 , 0.1667, 0.1429],
[ 0.25 , 0.2 , 0.1667, 0.1429, 0.125 ],
[ 0.2 , 0.1667, 0.1429, 0.125 , 0.1111]])
'''
n = 14
A = hilbert(n)
x = np.random.uniform(-10,10,n)
b = A @ x
A_inv = np.linalg.inv(A)
np.linalg.norm(np.eye(n) - A @ A_inv)
# 5.0516495470543212
np.linalg.cond(A)
# 2.2271635826494112e+17
A @ A_inv
'''
array([[ 1. , 0. , -0.0001, 0.0005, -0.0006, 0.0105, -0.0243,
0.1862, -0.6351, 2.2005, -0.8729, 0.8925, -0.0032, -0.0106],
[ 0. , 1. , -0. , 0. , 0.0035, 0.0097, -0.0408,
0.0773, -0.0524, 1.6926, -0.7776, -0.111 , -0.0403, -0.0184],
[ 0. , 0. , 1. , 0.0002, 0.0017, 0.0127, -0.0273,
0. , 0. , 1.4688, -0.5312, 0.2812, 0.0117, 0.0264],
[ 0. , 0. , -0. , 1.0005, 0.0013, 0.0098, -0.0225,
0.1555, -0.0168, 1.1571, -0.9656, -0.0391, 0.018 , -0.0259],
[-0. , 0. , -0. , 0.0007, 1.0001, 0.0154, 0.011 ,
-0.2319, 0.5651, -0.2017, 0.2933, -0.6565, 0.2835, -0.0482],
[ 0. , -0. , 0. , -0.0004, 0.0059, 0.9945, -0.0078,
-0.0018, -0.0066, 1.1839, -0.9919, 0.2144, -0.1866, 0.0187],
[-0. , 0. , -0. , 0.0009, -0.002 , 0.0266, 0.974 ,
-0.146 , 0.1883, -0.2966, 0.4267, -0.8857, 0.2265, -0.0453],
[ 0. , 0. , -0. , 0.0002, 0.0009, 0.0197, -0.0435,
1.1372, -0.0692, 0.7691, -1.233 , 0.1159, -0.1766, -0.0033],
[ 0. , 0. , -0. , 0.0002, 0. , -0.0018, -0.0136,
0.1332, 0.945 , 0.3652, -0.2478, -0.1682, 0.0756, -0.0212],
[ 0. , -0. , -0. , 0.0003, 0.0038, -0.0007, 0.0318,
-0.0738, 0.2245, 1.2023, -0.2623, -0.2783, 0.0486, -0.0093],
[-0. , 0. , -0. , 0.0004, -0.0006, 0.013 , -0.0415,
0.0292, -0.0371, 0.169 , 1.0715, -0.09 , 0.1668, -0.0197],
[ 0. , -0. , 0. , 0. , 0.0016, 0.0062, -0.0504,
0.1476, -0.2341, 0.8454, -0.7907, 1.4812, -0.15 , 0.0186],
[ 0. , -0. , 0. , -0.0001, 0.0022, 0.0034, -0.0296,
0.0944, -0.1833, 0.6901, -0.6526, 0.2556, 0.8563, 0.0128],
[ 0. , 0. , 0. , -0.0001, 0.0018, -0.0041, -0.0057,
-0.0374, -0.165 , 0.3968, -0.2264, -0.1538, -0.0076, 1.005 ]])
'''
row_names = ['Normal Eqns- Naive',
'QR Factorization',
'SVD',
'Scipy lstsq']
name2func = {'Normal Eqns- Naive': 'ls_naive',
'QR Factorization': 'ls_qr',
'SVD': 'ls_svd',
'Scipy lstsq': 'scipylstq'}
pd.options.display.float_format = '{:,.9f}'.format
df = pd.DataFrame(index=row_names, columns=['Time', 'Error'])
for name in row_names:
fcn = name2func[name]
t = timeit.timeit(fcn + '(A,b)', number=5, globals=globals())
coeffs = locals()[fcn](A, b)
df.set_value(name, 'Time', t)
df.set_value(name, 'Error', regr_metrics(b, A @ coeffs)[0])
SVD 在这里最好
不要重新运行。
df
| Time | Error | |
|---|---|---|
| Normal Eqns- Naive | 0.001334339 | 3.598901966 |
| QR Factorization | 0.002166139 | 0.000000000 |
| SVD | 0.001556937 | 0.000000000 |
| Scipy lstsq | 0.001871590 | 0.000000000 |
即使A是稀疏的, 通常是密集的。对于大型矩阵,
通常是密集的。对于大型矩阵, 放不进内存。
放不进内存。
运行时间
- 矩阵求逆:

- 矩阵乘法:

- Cholesky:

- QR,Gram Schmidt:
 ,
, (Trefethen 第 8 章)
(Trefethen 第 8 章) - QR,Householder:2
 (Trefethen 第 10 章)
(Trefethen 第 10 章) - 求解三角形系统:

为什么 Cholesky 较快:

QR 最优的一个案例
m=100
n=15
t=np.linspace(0, 1, m)
# 范德蒙矩阵
A=np.stack([t**i for i in range(n)], 1)
b=np.exp(np.sin(4*t))
# 这将使解决方案标准化为 1
b /= 2006.787453080206
from matplotlib import pyplot as plt
%matplotlib inline
plt.plot(t, b)
# [<matplotlib.lines.Line2D at 0x7fdfc1fa7eb8>]

检查我们得到了 1:
1 - ls_qr(A, b)[14]
# 1.4137685733217609e-07
不好的条件数:
kappa = np.linalg.cond(A); kappa
# 5.827807196683593e+17
row_names = ['Normal Eqns- Naive',
'QR Factorization',
'SVD',
'Scipy lstsq']
name2func = {'Normal Eqns- Naive': 'ls_naive',
'QR Factorization': 'ls_qr',
'SVD': 'ls_svd',
'Scipy lstsq': 'scipylstq'}
pd.options.display.float_format = '{:,.9f}'.format
df = pd.DataFrame(index=row_names, columns=['Time', 'Error'])
for name in row_names:
fcn = name2func[name]
t = timeit.timeit(fcn + '(A,b)', number=5, globals=globals())
coeffs = locals()[fcn](A, b)
df.set_value(name, 'Time', t)
df.set_value(name, 'Error', np.abs(1 - coeffs[-1]))
df
| Time | Error | |
|---|---|---|
| Normal Eqns- Naive | 0.001565099 | 1.357066025 |
| QR Factorization | 0.002632104 | 0.000000116 |
| SVD | 0.003503785 | 0.000000116 |
| Scipy lstsq | 0.002763502 | 0.000000116 |
通过正规方程求解最小二乘的解决方案通常是不稳定的,尽管对于小条件数的问题是稳定的。
低秩
m = 100
n = 10
x = np.random.uniform(-10,10,n)
A2 = np.random.uniform(-40,40, [m, int(n/2)]) # removed np.asfortranarray
A = np.hstack([A2, A2])
A.shape, A2.shape
# ((100, 10), (100, 5))
b = A @ x + np.random.normal(0,1,m)
row_names = ['Normal Eqns- Naive',
'QR Factorization',
'SVD',
'Scipy lstsq']
name2func = {'Normal Eqns- Naive': 'ls_naive',
'QR Factorization': 'ls_qr',
'SVD': 'ls_svd',
'Scipy lstsq': 'scipylstq'}
pd.options.display.float_format = '{:,.9f}'.format
df = pd.DataFrame(index=row_names, columns=['Time', 'Error'])
for name in row_names:
fcn = name2func[name]
t = timeit.timeit(fcn + '(A,b)', number=5, globals=globals())
coeffs = locals()[fcn](A, b)
df.set_value(name, 'Time', t)
df.set_value(name, 'Error', regr_metrics(b, A @ coeffs)[0])
df
| Time | Error | |
|---|---|---|
| Normal Eqns- Naive | 0.001227640 | 300.658979382 |
| QR Factorization | 0.002315920 | 0.876019803 |
| SVD | 0.001745647 | 1.584746056 |
| Scipy lstsq | 0.002067989 | 0.804750398 |
比较
比较我们的结果和上面:
df
| # rows | 100 | 1000 | 10000 | ||||||
|---|---|---|---|---|---|---|---|---|---|
| # cols | 20 | 100 | 1000 | 20 | 100 | 1000 | 20 | 100 | 1000 |
| Normal Eqns- Naive | 0.001276 | 0.003634 | NaN | 0.000960 | 0.005172 | 0.293126 | 0.002226 | 0.021248 | 1.164655 |
| Normal Eqns- Cholesky | 0.001660 | 0.003958 | NaN | 0.001665 | 0.004007 | 0.093696 | 0.001928 | 0.010456 | 0.399464 |
| QR Factorization | 0.002174 | 0.006486 | NaN | 0.004235 | 0.017773 | 0.213232 | 0.019229 | 0.116122 | 2.208129 |
| SVD | 0.003880 | 0.021737 | NaN | 0.004672 | 0.026950 | 1.280490 | 0.018138 | 0.130652 | 3.433003 |
| Scipy lstsq | 0.004338 | 0.020198 | NaN | 0.004320 | 0.021199 | 1.083804 | 0.012200 | 0.088467 | 2.134780 |
来自 Trefethen(第 84 页):
正规方程式/ Cholesky 在生效时速度最快。 Cholesky 只能用于对称正定矩阵。 此外,对于具有高条件数或具有低秩的矩阵,正规方程/ Cholesky 是不稳定的。
数值分析师推荐通过 QR 进行线性回归,作为多年的标准方法。 它自然,优雅,适合“日常使用”。