十六、降维
译者:飞龙
降维是将数据集转换为较低维空间的过程。
有关降维的解释,以及实际数据中的示例,请访问这里。
降维的概述
数据集可能非常大,不仅包含数百万个数据点,还包含有关每个数据点的大量特征。 我们可以将数据集的每个特征都视为“维度”。 对于高维数据,我们可能希望或需要尝试减少这些维度。
最简单的方法可能只是放弃一些维度,我们甚至可以选择放弃看起来可能最不实用的维度。这将是一种简单的降维方法。
但通常情况下,我们希望以保留最多信息的方式减少维度的数量。 一种方法可能是聚类 - 如果我们对高维数据进行聚类分析,我们可以重新编码数据,来通过它的簇标签存储每个点,从而可能在更少的维度中维护更多信息。
在这里,我们将探索另一种降维的常用方法 - 主成分分析(PCA)。
主成分分析(PCA)是将数据集转换为主成分的过程,按捕获了多少方差排序。
有关涵盖 PCA 完整教程的论文,请访问这里。更多技术概述和解释,请查看这个博文。
主成分分析
要使用 PCA 进行降维,我们可以选择保留n个成分,其中n是一个低于数据集原始维数的数字。
通过使用 PCA 转换数据并保留n个成分,我们能够在较低维空间中保持原始数据的最大方差。
最广泛地说,PCA 试图利用变量的相关结构,并使用这种结构对数据进行编码。
例如,如果我们数据的特征x1和x2是相关的,那么 PCA 会查找如何将数据重新组织成一些新的维度x_pc,它捕获了两者之间的大部分共享方差(相关结构)。
在这个笔记本中,我们将遵循一个简单的 PCA 示例,使用 PCA 将数据从 2D 转换为 1D。
实际上,从m D 转换为 n D 数据,PCA 最有用,其中m非常大,而n < m。
# 导入
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
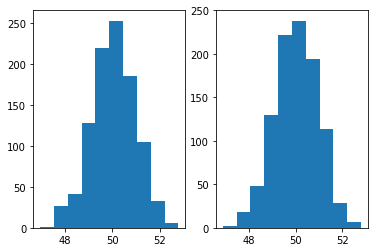
## 创建一些随机的互相关数据
# 设置
means = [50, 50]
covs = [[1, .75], [.75, 1]]
n = 1000
# 生成数据
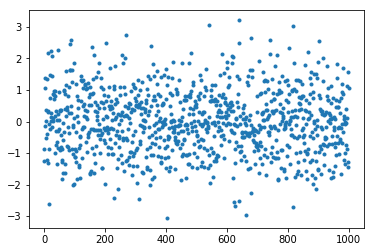
dat = np.random.multivariate_normal(means, covs, n)
# 绘制我们的两个随机变量
f, ax = plt.subplots(1, 2)
ax[0].hist(dat[:, 0]);
ax[1].hist(dat[:, 1]);
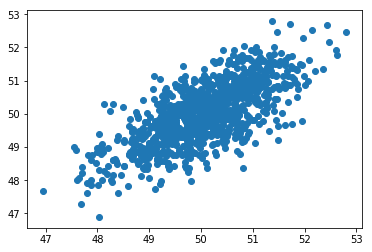
# 查看数据如何相互关联
plt.scatter(dat[:, 0], dat[:, 1])
# <matplotlib.collections.PathCollection at 0x1117902b0>
任务
假设我们想要或需要数据的较低维度表示来进行进一步分析。
虽然降维不是简单地选择维度的子集,而是在较小数量的维度中尝试从原始空间捕获最多信息的过程。
注意:这里我们将从 2D -> 1D 开始,为了简化和方便。实际上,当你有一个难以处理的大量维度,比如说 20,000,并希望将数据转换为一个较低维度的空间(捕获尽可能多的信息)时,它是最有用的,但是或许包含接近 20 个维度,可能便于管理。
在这里,我们将使用主成分分析(PCA)来降维。
# 初始化PCA模型,此处指定 1 个成分
pca = PCA(n_components=1, whiten=True)
# 拟合 PCA 模型,并应用它来转换数据
out = pca.fit_transform(dat)
# 在新空间中查看转换后的数据
# 注意:由于新数据是 1D,因此这里的 x 轴只是索引号
plt.plot(out, '.')
# [<matplotlib.lines.Line2D at 0x1118b8ef0>]